
En este post describimos paso a paso un ejercicio de ciencia de datos en el que tratamos de entrenar un modelo de deep learning con el objetivo de clasificar automáticamente imágenes médicas de personas sanas y enfermas.
El diagnóstico por radio-imagen existe desde hace muchos años en los hospitales de los países desarrollados, sin embargo, siempre ha existido una fuerte dependencia de personal altamente especializado. Desde el técnico que opera los instrumentos hasta el médico radiólogo que interpreta las imágenes. Con nuestras capacidades analíticas actuales, somos capaces de extraer medidas numéricas como el volumen, la dimensión, la forma y la tasa de crecimiento (entre otras) a partir del análisis de imagen. A lo largo de este post trataremos de explicarte, mediante un sencillo ejemplo, la potencia de los modelos de inteligencia artificial para ampliar las capacidades humanas en el campo de la medicina.
Este post explica el ejercicio práctico (sección Action) asociado al informe “Tecnologías emergentes y datos abiertos: introducción a la ciencia de datos aplicada al análisis de imagen”. Dicho informe introduce los conceptos fundamentales que permiten comprender cómo funciona el análisis de imagen, detallando los principales casos de aplicación en diversos sectores y resaltando el papel de los datos abiertos en su ejecución.
Proyectos previos
No podríamos haber preparado este ejercicio sin el trabajo y el esfuerzo previo de otros entusiastas de la ciencia de datos. A continuación, te dejamos una pequeña nota y las referencias a estos trabajos previos.
- Este ejercicio es una adaptación del proyecto original de Michael Blum sobre el desafío STOIC2021 - dissease-19 AI challenge. El proyecto original de Michael, partía de un conjunto de imágenes de pacientes con patología Covid-19, junto con otros pacientes sanos para hacer contraste.
- En una segunda aproximación, Olivier Gimenez utilizó un conjunto de datos similar al del proyecto original publicado en una competición de Kaggle. Este nuevo dataset (250 MB) era considerablemente más manejable que el original (280GB). El nuevo dataset contenía algo más de 1000 imágenes de pacientes sanos y enfermos. El código del proyecto de Olivier puede encontrarse en el siguiente repositorio.
Conjuntos de datos
En nuestro caso, inspirándonos en estos dos fantásticos proyectos previos, hemos construido un ejercicio didáctico apoyándonos en una serie de herramientas que facilitan la ejecución del código y la posibilidad de examinar los resultados de forma sencilla. El conjunto de datos original (de rayos-x de tórax) comprende 112.120 imágenes de rayos-X (vista frontal) de 30.805 pacientes únicos. Las imágenes se acompañan con las etiquetas asociadas de catorce enfermedades (donde cada imagen puede tener múltiples etiquetas), extraídas de los informes radiológicos asociados utilizando procesamiento de lenguaje natural (NLP). Partiendo del conjunto de imágenes médicas original hemos extraído (utilizando algunos scripts) una muestra más pequeña y acotada (tan solo personas sanas frente a personas con una sola patología) para facilitar este ejercicio. En particular, la patología escogida es el neumotórax.
Si quieres ampliar la información sobre el campo de procesamiento del lenguaje natural puedes consultar el siguiente informe que ya publicamos en su momento. Además, en el post 10 repositorios de datos públicos relacionados con la salud y el bienestar se cita al NIH como un ejemplo de fuente de datos sanitarios de calidad. En particular, nuestro conjunto de datos está disponible públicamente aquí.
Herramientas
Para la realización del tratamiento previo de los datos (entorno de trabajo, programación y redacción del mismo) se ha utilizado R (versión 4.1.2) y RStudio (2022-02-3). Los pequeños scripts de ayuda a la descarga y ordenación de ficheros se han escrito en Python 3.
Acompañando a este post, hemos creado un cuaderno de Jupyter con el que poder experimentar de forma interactiva a través de los diferentes fragmentos de código que van desarrollando nuestro ejemplo. El objetivo de este ejercicio es entrenar a un algoritmo para que sea capaz de clasificar automáticamente una imagen de una radiografía de pecho en dos categorías (persona enferma vs persona no-enferma). Para facilitar la ejecución del ejercicio por parte de los lectores que así lo deseen, hemos preparado el cuaderno de Jupyter en el entorno de Google Colab que contiene todos los elementos necesarios para reproducir el ejercicio paso a paso. Google Colab o Collaboratory es una herramienta gratuita de Google que te permite programar y ejecutar código en Python (y también en R) sin necesidad de instalar ningún software adicional. Es un servicio online y para usarlo tan solo necesitas tener una cuenta de Google.
Flujo lógico del análisis de datos
Nuestro cuaderno de Jupyter, realiza las siguientes actividades diferenciadas que podrás seguir en el propio documento interactivo cuándo lo vayas ejecutando sobre Google Colab.
- Instalación y carga de dependencias.
- Configuración del entorno de trabajo
- Descarga, carga y pre-procesamiento de datos necesarios (imágenes médicas) en el entorno de trabajo.
- Pre-visualización de las imágenes cargadas.
- Preparación de los datos para entrenamiento del algoritmo.
- Entrenamiento del modelo y resultados.
- Conclusiones del ejercicio.
A continuación, hacemos un repaso didáctico del ejercicio, enfocando nuestras explicaciones en aquellas actividades que son más relevantes respecto al ejercicio de análisis de datos:
- Descripción del análisis de datos y entrenamiento del modelo
- Modelización: creación del conjunto de imágenes de entrenamiento y entrenamiento del modelo
- Analisis del resultado del entrenamiento
- Conclusiones
Descripción del análisis de datos y entrenamiento del modelo
Los primeros pasos que encontraremos recorriendo el cuaderno de Jupyter son las actividades previas al análisis de imágenes propiamente dicho. Como en todos los procesos de análisis de datos, es necesario preparar el entorno de trabajo y cargar las librerías necesarias (dependencias) para ejecutar las diferentes funciones de análisis. El paquete de R más representativo de este conjunto de dependencias es Keras. En este artículo ya comentamos sobre el uso de Keras como framework de Deep Learning. Adicionalmente también son necesarios los siguientes paquetes: httr; tidyverse; reshape2;patchwork.
A continuación, debemos descargar a nuestro entorno el conjunto de imágenes (datos) con el que vamos a trabajar. Como hemos comentado previamente, las imágenes se encuentran en un almacenamiento remoto y solo las descargamos en Colab en el momento de analizarlas. Tras ejecutar las secciones de código que descargan y descomprimen los ficheros de trabajo que contienen las imágenes médicas encontraremos dos carpetas (No-finding y Pneumothorax) que contienen los datos de trabajo.

Una vez que disponemos de los datos de trabajo en Colab, debemos cargarlas en la memoria del entorno de ejecución. Para ello hemos creado una función que verás en el cuaderno denominada process_pix(). Esta función, va a buscar las imágenes a las carpetas anteriores y las carga en memoria, además de pasarlas a escala de grises y normalizarlas todas a un tamaño de 100x100 pixels. Para no exceder los recursos que nos proporciona de forma gratuita Google Colab, limitamos la cantidad de imágenes que cargamos en memoria a 1000 unidades. Es decir, el algoritmo va a ser entrenado con 1000 imágenes, entre las que va usar para entrenamiento y las que va a usar para la validación posterior.
Una vez tenemos las imágenes perfectamente clasificadas, formateadas y cargadas en memoria hacemos una visualización rápida para verificar que son correctas.Obtenemos los siguientes resultados:

Obviamente, a ojos de un observador no experto no se ven diferencias significativas que nos permitan extraer ninguna conclusión. En los siguientes pasos veremos cómo el modelo de inteligencia artificial sí que tiene mejor ojo clínico que nosotros.
Modelización
Creación del conjunto de imágenes de entrenamiento
Como comentamos en los pasos previos, disponemos de un conjunto de 1000 imágenes de partida cargadas en el entorno de trabajo. Hasta este momento, tenemos clasificadas (por un especialista en rayos-x) aquellas imágenes de pacientes que presentan indicios de neumotórax (en la ruta "./data/Pneumothorax") y aquellos pacientes sanos (en la ruta "./data/No-Finding")
El objetivo de este ejercicio es, justamente, demostrar la capacidad de un algoritmo de asistir al especialista en la clasificación (o detección de signos de enfermedad en la imagen de rayos-x). Para esto, tenemos que mezclar las imágenes, para conseguir un conjunto homogéneo que el algoritmo tendrá que analizar y clasificar valiéndose solo de sus características. El siguiente fragmento de código, asocia un identificador (1 para personas enfermas y 0 para personas sanas) para, posteriormente, tras el proceso de clasificación del algoritmo, poder verificar aquellas que el modelo ha clasificado de forma correcta o incorrecta.

Bien, ahora tenemos un conjunto “df” uniforme de 1000 imágenes mezcladas con pacientes sanos y enfermos. A continuación, dividimos en dos este conjunto original. El 80% del conjunto original, lo vamos a utilizar para entrenar el modelo. Esto es, el algoritmo utilizará las características de las imágenes para crear un modelo que permita concluir si una imagen se corresponde con el identificador 1 o 0. Por otro lado, el 20% restante de la mezcla homogénea la vamos a utilizar para comprobar si el modelo, una vez entrenado, es capaz de tomar una imagen cualquiera y asignarle el 1 o el 0 (enfermo, no enfermo).

Entrenamiento del modelo
Listo, solo nos queda configurar el modelo y entrenar con el anterior conjunto de datos.
Antes de entrenar, veréis unos fragmentos de código que sirven para configurar el modelo que vamos a entrenar. El modelo que vamos a entrenar es de tipo clasificador binario. Esto significa que es un modelo que es capaz de clasificar los datos (en nuestro caso imágenes) en dos categorías (en nuestro caso sano o enfermo). El modelo escogido se denomina CNN o Convolutional Neural Network. Su propio nombre ya nos indica que es un modelo de redes neuronales y por lo tanto cae dentro de la disciplina de Deep Learning o aprendizaje profundo. Estos modelos se basan en capas de características de los datos que se van haciendo más profundas a medida que la complejidad del modelo aumenta. Os recordamos que el término deep hace referencia, justamente, a la profundidad del número de capas mediante las cuales estos modelos aprenden.
Nota: los siguientes fragmentos de código son los más técnicos del post. La documentación introductoria se puede encontrar aquí, mientras que toda la documentación técnica sobre las funciones del modelo está accesible aquí.
Finalmente, tras la configuración del modelo, estamos en disposición de entrenar el modelo. Como comentamos, entrenamos con el 80% de las imágenes y validamos el resultado con el 20% restante.

Resultado del entrenamiento
Bien, ya hemos entrenado nuestro modelo. ¿Y ahora qué? Las siguientes gráficas nos proporcionan una visualización rápida sobre cómo se comporta el modelo sobre las imágenes que hemos reservado para validar. Básicamente, estas figuras vienen a representar (la del panel inferior) la capacidad del modelo de predecir la presencia (identificador 1) o ausencia (identificador 0) de enfermedad (en nuestro caso neumotórax). La conclusión es que cuando el modelo entrenado con las imágenes de entrenamiento (aquellas de las que se sabe el resultado 1 o 0) se aplica al 20% de las imágenes de las cuales no se sabe el resultado, el modelo acierta aproximadamente en el 85% (0.87309) de las ocasiones.

En efecto, cuando solicitamos la evaluación del modelo para saber qué tan bien clasifica enfermedades el resultado nos indica la capacidad de nuestro modelo recién entrenado para clasificar de forma correcta el 0.87309 de las imágenes de validación.
Hacemos ahora algunas predicciones sobre imágenes de pacientes. Es decir, una vez entrenado y validado el modelo, nos preguntamos cómo va a clasificar las imágenes que le vamos a dar ahora. Como sabemos "la verdad" (lo que se denomina el ground truth) sobre las imágenes, comparamos el resultado de la predicción con la verdad. Para comprobar los resultados de la predicción (que variarán en función del número de imágenes que se usen en el entrenamiento) se utiliza lo que en ciencia de datos se denomina la matriz de confusión. La matriz de confusión:
- coloca en la posición (1,1) los casos que SÍ tenían enfermedad y el modelo clasifica como "con enfermedad"
- coloca en la posición (2,2), los casos que NO tenían enfermedad y el modelo clasifica como "sin enfermedad"
Es decir, estas son las posiciones en las que el modelo "acierta" en su clasificación.
En las posiciones contrarias, es decir, la (1,2) y la (2,1) son las posiciones en las que el modelo se "equivoca". Así, la posición (1,2) son los resultados que el modelo clasifica como CON enfermedad y la realidad es que eran pacientes sanos. La posición (2,1), justo lo contrario.
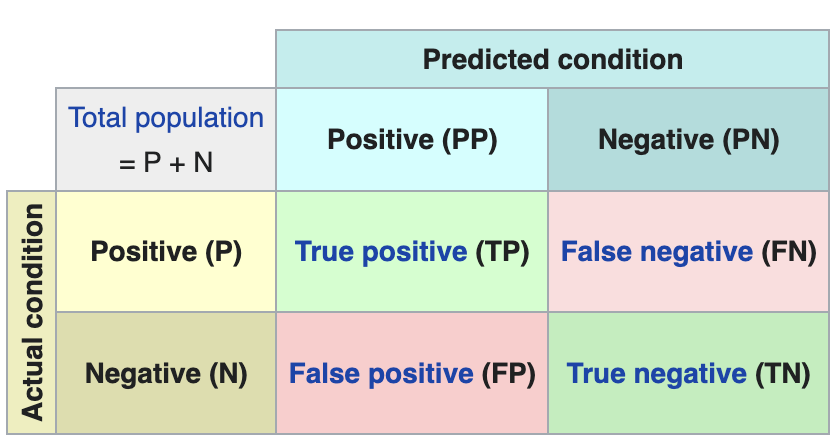
Ejemplo explicativo sobre cómo funciona la matriz de confusión. Fuente: wikipedia https://en.wikipedia.org/wiki/Confusion_matrix
En nuestro ejercicio, el modelo nos proporciona los siguientes resultados:


Es decir, 81 pacientes tenían esta enfermedad y el modelo los clasifica de forma correcta. De la misma forma, 91 pacientes estaban sanos y el modelo los clasifica, igualmente, de forma correcta. Sin embargo, el modelo clasifica cómo enfermos, 13 pacientes que estaban sanos. Y al contrario, el modelo clasifica cómo sanos a 12 pacientes que en realidad estaban enfermos. Cuando sumamos los aciertos del modelo 81+91 y lo dividimos sobre la muestra de validación total obtenemos el 87% de precisión del modelo.
Conclusiones
En este post te hemos guiado a través de un ejercicio didáctico que consiste en entrenar un modelo de inteligencia artificial para realizar clasificaciones de imágenes de radiografías de pecho con el objetivo de determinar automáticamente si una persona está enferma o sana. Por sencillez, hemos escogido pacientes sanos y pacientes que presentan un neumotórax (solo dos categorías) previamente diagnosticados por un médico. El viaje que hemos realizado nos ofrece una idea de las actividades y las tecnologías involucradas en el análisis automatizado de imágenes mediante inteligencia artificial. El resultado del entrenamiento nos ofrece un sistema de clasificación razonable para el screening automático con un 87% de precisión en sus resultados. Los algoritmos y las tecnologías avanzadas de análisis de imagen son y cada vez más serán, un complemento indispensable en múltiples ámbitos y sectores como, por ejemplo, la medicina. En los próximos años veremos cómo se consolidan los sistemas que, de forma natural, combinan habilidades de humanos y máquinas en procesos costosos, complejos o peligrosos. Los médicos y otros trabajadores verán aumentadas y reforzadas sus capacidades gracias a la inteligencia artificial. La combinación de fuerzas entre máquinas y humanos nos permitirá alcanzar cotas de precisión y eficiencia nunca vistas hasta la fecha. Esperamos que con este ejercicio os hayamos ayudado a entender un poco más cómo funcionan estas tecnologías. No olvides completar tu aprendizaje con el resto de materiales que acompañan este post.
Contenido elaborado por Alejandro Alija, experto en Transformación Digital. Los contenidos y los puntos de vista reflejados en esta publicación son responsabilidad exclusiva de su autor.