Los datos abiertos de salud son uno de los activos más valiosos de nuestra sociedad. Bien gestionados y compartidos de forma responsable, pueden salvar vidas, impulsar descubrimientos médicos o incluso optimizar recursos hospitalarios. Sin embargo, durante décadas, estos datos han permanecido fragmentados en silos institucionales, con formatos incompatibles y barreras técnicas y legales que dificultaban su reutilización. Ahora, la Unión Europea está cambiando radicalmente el panorama con una estrategia ambiciosa que combina dos enfoques complementarios:
- Facilitar el acceso abierto a estadísticas y datos agregados no sensibles.
- Crear infraestructuras seguras para compartir datos personales de salud bajo estrictas garantías de privacidad.
En España, esta transformación ya está en marcha a través del Espacio Nacional de Datos de Salud o grupos de investigación que están a la vanguardia en el uso innovador de datos de salud. Iniciativas como IMPACT-Data, que integra datos médicos para impulsar la medicina de precisión, demuestran el potencial de trabajar con datos de salud de manera estructurada y segura. Y para facilitar que todos estos datos sean fáciles de encontrar y reutilizar se implementan estándares como HealthDCAT-AP.
Todo ello está perfectamente alineado con la estrategia europea del Reglamento del Espacio Europeo de Datos de Salud (EHDS), publicado oficialmente en marzo de 2025 que se integra también con la Directiva de Datos Abiertos (ODD), en vigor desde 2019. Aunque ambos marcos regulatorios tienen alcances distintos, su interacción ofrece oportunidades extraordinarias para la innovación, la investigación y la mejora de la atención sanitaria en toda Europa.
Un reciente informe elaborado por Capgemini Invent para data.europa.eu analiza estas sinergias. En este post, exploramos las principales conclusiones de este trabajo y reflexionamos sobre su relevancia para el ecosistema español de datos abiertos.
Dos marcos complementarios para un objetivo común
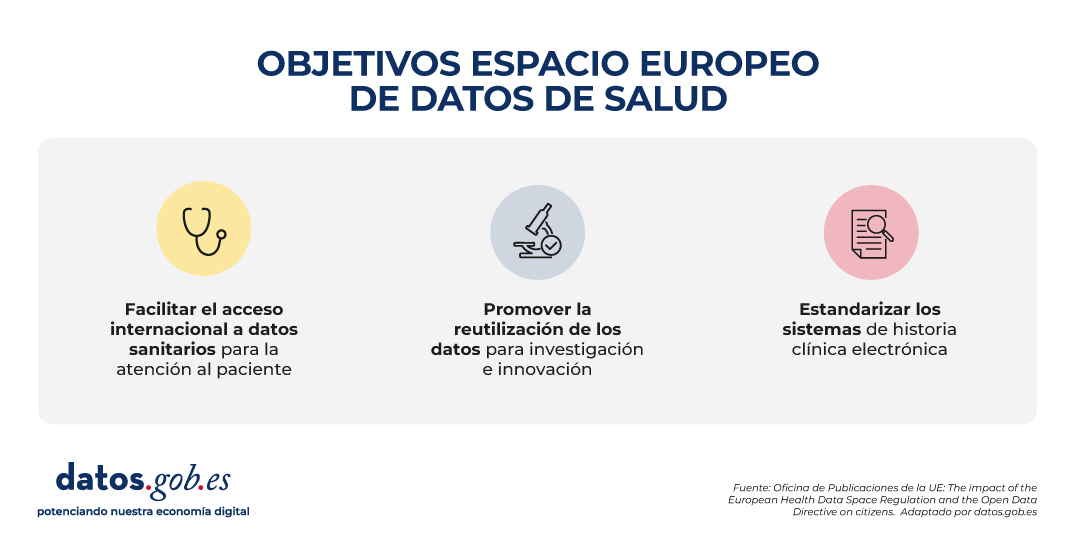
Por un lado, el Espacio Europeo de Datos de Salud se centra específicamente en datos de salud y persigue tres objetivos fundamentales:
- Facilitar el acceso internacional a datos sanitarios para la atención al paciente (uso primario).
- Promover la reutilización de estos datos para investigación, políticas públicas e innovación (uso secundario).
- Estandarizar técnicamente los sistemas de historia clínica electrónica (HCE) para mejorar la interoperabilidad transfronteriza.
Por su parte, la Directiva de Datos Abiertos tiene un alcance más amplio: promueve que el sector público ponga a disposición de cualquier usuario datos gubernamentales para su reutilización libre. Esto incluye los conjuntos de datos de alto valor (High-Value Datasets) que deben publicarse gratuitamente, en formatos legibles por máquina y a través de API en seis categorías entre las que no se encontraba “salud” originalmente. Sin embargo, en la propuesta de ampliación de las nuevas categorías que publicó la UE sí aparece la categoría de salud.
La complementariedad entre ambos marcos regulatorios es evidente: mientras la ODD facilita el acceso abierto a estadísticas sanitarias agregadas y no sensibles, el EHDS regula el acceso controlado a datos individuales de salud bajo condiciones estrictas de seguridad, consentimiento y gobernanza. Juntos, conforman un sistema escalonado de compartición de datos que maximiza su valor social sin comprometer la privacidad, en total cumplimiento con el Reglamento General de Protección de Datos (RGPD).
Principales beneficios ordenador por grupos de usuarios
El informe analiza cuatro grupos de usuarios principales y examina tanto los beneficios potenciales como los desafíos que enfrentan al combinar datos del EHDS con datos abiertos.
-
Pacientes: empoderamiento informado con barreras prácticas
Los pacientes europeos obtendrán acceso más rápido y seguro a sus propias historias clínicas electrónicas, especialmente en contextos transfronterizos gracias a infraestructuras como MyHealth@EU. Este proyecto resulta especialmente útil para ciudadanos europeos que se encuentren desplazados en otro país europeo. .
Otro proyecto interesante que informa a la ciudadanía es PatientsLikeMe que reúne a más 850.000 pacientes con enfermedades raras o crónicas en una comunidad online que comparte información de interés sobre tratamientos y otras cuestiones.
-
Profesionales de la salud potencial subordinado a la integración
Por otro lado, los profesionales sanitarios podrán acceder antes y de manera más sencilla a datos clínicos de pacientes, incluso a través de fronteras, mejorando la continuidad asistencial y la calidad del diagnóstico y tratamiento.
La combinación con datos abiertos podría amplificar estos beneficios si se desarrollan herramientas que integren ambas fuentes de información directamente en los sistemas de historia clínica electrónica.
3. Responsables políticos: datos para mejores decisiones
Los cargos públicos son beneficiarios naturales de la convergencia entre EHDS y datos abiertos. La posibilidad de combinar datos salud detallados (previa solicitud y autorización a través de los Organismos de Acceso a Datos Sanitarios que cada Estado miembro debe establecer) con información estadística y contextual abierta permitiría desarrollar políticas basadas en evidencia mucho más sólida.
El informe menciona casos de uso como la combinación de datos de salud con información medioambiental para evaluar impactos sanitarios. Un ejemplo real es el proyecto francés Green Data for Health, que cruza datos abiertos sobre contaminación acústica con información sobre prescripciones de medicamentos para el sueño de más de 10 millones de habitantes, investigando correlaciones entre ruido ambiental y trastornos del sueño.
4. Investigadores y reutilizadores: los principales beneficiarios inmediatos
Los investigadores, académicos e innovadores constituyen el grupo que más directamente se beneficiará de la sinergia EHDS-ODD ya que disponen de las habilidades y herramientas necesarias para localizar, acceder, combinar y analizar datos de múltiples fuentes. Además, su trabajo ya implica habitualmente la integración de diversos conjuntos de datos.
Un estudio reciente publicado en PLOS Digital Health sobre el caso de Andalucía demuestra cómo los datos abiertos en salud pueden democratizar la investigación en IA sanitaria y mejorar la equidad en el tratamiento.
El desarrollo del EHDS está siendo apoyado por programas europeos como EU4Health, Horizon Europe y proyectos específicos como TEHDAS2, que ayudan a definir estándares técnicos y pilotar aplicaciones reales.
Recomendaciones para maximizar el impacto
El informe concluye con cuatro recomendaciones clave que resultan particularmente relevantes para el ecosistema español de datos abiertos:
- Estimular la investigación en la intersección EHDS-datos abiertos mediante financiación específica. Es fundamental incentivar que los investigadores que combinan estas fuentes traduzcan sus hallazgos en aplicaciones prácticas: protocolos clínicos mejorados, herramientas de decisión, estándares de calidad actualizados.
- Evaluar y facilitar el uso directo por profesionales y pacientes. Promover la alfabetización en datos y desarrollar aplicaciones intuitivas integradas en los sistemas existentes (como las historias clínicas electrónicas) podría cambiar esta situación.
- Fortalecer la gobernanza mediante educación y marcos regulatorios claros. A medida que se vayan operativizando las entidades técnicas del EHDS , será esencial contar con una regulación clara que defina unos marcos regulatorios comunes..
- Monitorizar, evaluar y adaptar. El período 2025-2031 verá la entrada en vigor gradual de los distintos requisitos del EHDS. Se recomienda realizar evaluaciones periódicas para valorar cómo se está utilizando realmente el EHDS, qué combinaciones con datos abiertos están generando más valor, y qué ajustes son necesarios.
Además, para que todo esto funcione, el informe sugiere que portales como data.europa.eu (y por extensión, datos.gob.es) deberían destacar ejemplos prácticos que demuestren cómo se complementan los datos abiertos con los datos protegidos de espacios sectoriales, inspirando así nuevas aplicaciones.
En general, el papel de los portales de datos abiertos será fundamental en este ecosistema emergente: no solo como proveedores de conjuntos de datos de calidad, sino también como facilitadores de conocimiento, espacios de encuentro entre comunidades y catalizadores de innovación. El futuro de la sanidad europea se está escribiendo ahora, y los datos abiertos tienen un papel protagonista en esa historia.
En la era digital, los avances tecnológicos han transformado el sector de la investigación médica. Uno de los factores que contribuyen al desarrollo tecnológico en este ámbito son los datos y, en especial, los datos abiertos. La apertura y disponibilidad de la información que se obtiene de investigaciones sanitarias aporta múltiples beneficios a la comunidad científica. Los datos abiertos en el sector salud fomentan la colaboración entre investigadores, aceleran el proceso de validación de resultados en estudios y, en definitiva, ayudan a salvar vidas.
La relevancia de este tipo de datos también se manifiesta en la intención prioritaria de constituir el proyecto de espacio europeo de datos sanitarios (EEDS), el primer espacio común de datos de la UE que surge de la Estrategia Europea de Datos y una de las prioridades de la Comisión para el período 2019-2025. Tal y como plantea la Comisión Europea en su propuesta, el EEDS este espacio contribuirá a promover un mejor intercambio y acceso a diferentes tipos de datos sanitarios, no solo para apoyar la prestación de asistencia médica sino también para la investigación sanitaria y la elaboración de políticas en el ámbito de la salud.
Sin embargo, el tratamiento de este tipo de datos debe de ser adecuado, debido a la información sensible que albergan. Los datos personales relativos a la salud están considerados como una categoría especial por la Agencia Española de Protección de Datos (AEPD) y una brecha de datos personales, especialmente, en el sector de la salud, tiene un alto impacto personal y social.
Para evitar estos riesgos, los datos médicos se pueden anonimizar garantizando el cumplimiento normativo y de los derechos fundamentales y, así, proteger la privacidad de los pacientes. La Guía básica de anonimización elaborada por la AEPD a partir de la Personal Data Protection Commission Singapore (PDPC) define los conceptos clave de un proceso de anonimización, incluyendo términos, principios metodológicos, tipos de riesgos y técnicas existentes.
Una vez se realiza ese proceso, los datos médicos pueden contribuir a la investigación sobre enfermedades, lo que se traduce en mejoras en la eficacia de tratamientos y en el desarrollo de tecnologías de asistencia médica. Además, los datos abiertos en el sector salud permiten que los científicos compartan información, resultados y hallazgos de manera rápida y accesible, fomentando así la colaboración y la replicabilidad de los estudios.
En este sentido, existen diversas instituciones que comparten sus datos anonimizados para contribuir a la investigación sanitaria y el desarrollo de la ciencia. Una de ellas es la Fundación FISABIO (Fundación para el Fomento de la Investigación Sanitaria y Biomédica de la Comunitat Valenciana) que se ha convertido en un referente en el campo de la medicina gracias a su compromiso con la apertura y compartición de datos médicos. Como parte de esta institución, ubicada en la Comunidad Valenciana, existe la Unidad Mixta de Imagen Biomédica de FISABIO y la Fundación Príncipe Felipe (FISABIO-CIPF) que se dedica, entre otras tareas, al estudio y desarrollo de técnicas avanzadas de imagen médica para mejorar el diagnóstico y tratamiento de enfermedades.
Este grupo de investigación ha desarrollado diferentes proyectos sobre análisis de imagen médica. El resultado de todo su trabajo se publica bajo licencias de código abierto: desde el resultado de sus investigaciones hasta los repositorios de datos que emplean para entrenar modelos de inteligencia artificial y machine learning.
Para proteger los datos sensibles de los pacientes, también han desarrollado sus propias técnicas de anonimización y seudonimización de imágenes e informes médicos mediante un modelo de Procesamiento del Lenguaje Natural (NLP) por el que los datos anonimizados se pueden sustituir por valores sintéticos. Siguiendo su técnica, se puede borrar la información facial de resonancias magnéticas cerebrales empleando un software libre de deep learning.
BIMCV: Banco de imágenes médicas de la Comunidad Valenciana
Uno de los mayores hitos de la Conselleria de Sanidad Universal y Salud Pública, a través de la Fundación y el hospital San Juan de Alicante, es la creación y mantenimiento del Banco de Imágenes Médicas de la Comunidad Valenciana, BIMCV (por sus siglas en inglés, Medical Imaging Databank of the Valencia Region), un repositorio de conocimiento para lograr “avances tecnológicos en imágenes médicas y proporcionar servicios de cobertura tecnológica para apoyar proyectos de I+D”, tal y como explican en su web.
BIMCV se aloja en XNAT, una plataforma que contiene imágenes de código abierto para la investigación basada en imágenes, y que es accesible bajo previo registro y/o bajo demanda. Actualmente, el Banco de Imágenes Médicas de la Comunidad Valenciana incluye datos abiertos procedentes de investigaciones realizada en diversos centros sanitarios de la región: alberga datos de más de 90.000 sujetos recogidos en más de 150.000 sesiones.
Nuevo conjunto de datos de imágenes radiológicas
Recientemente, la Unidad Mixta de Imagen Biomédica de FISABIO y la Fundación Príncipe Felipe (FISABIO-CIPF) ha publicado en abierto la tercera y última iteración de datos del proyecto BIMCV-COVID-19: iniciativa con la que liberaron datos de imagen de radiologías de tórax realizadas a pacientes con COVID-19, así como los modelos que habían entrenado para detección de diferentes patologías de Rx tórax, gracias al apoyo de la Conselleria de Innovación, la Conselleria de Sanidad y los Fondos de la Unión Europea REACT-UE. Todo ello, “para que pueda ser utilizado por empresas del sector o simplemente para investigación”, explica María de la Iglesia, directora de la Unidad. “Creemos que la reproducibilidad es de gran relevancia e importancia en el sector salud", añade. Los conjuntos de datos y el resultado de sus investigaciones se pueden consultar aquí.
Los hallazgos están mapeados en terminología estándar del Sistema Unificado de Lenguaje Médico (UMLS) (como propuesta de los resultados de la tesis doctoral de la Oncóloga e Ingeniera Informática Dra. Aurelia Bustos)y almacenados en alta resolución con etiquetas anatómicas en un formato de Estructura de Datos de Imágenes Médicas (MIDS). Entre la información almacenada, se encuentran datos demográficos del paciente, el tipo de proyección y los parámetros de adquisición del estudio de imagen, entre otros, todo ello anonimizado.
La contribución que este tipo de proyectos sobre datos abiertos aportan a la sociedad, no solo beneficia a los investigadores y profesionales de la salud, sino que también permite el desarrollo de soluciones que pueden tener un impacto relevante en la mejora de la atención médica. Una de ellas puede ser la IA generativa que proporciona interesantes resultados que los profesionales sanitarios, priorizando su criterio, pueden tomar en consideración para personalizar el diagnóstico y proponer un tratamiento más eficaz.
Por otro lado, la digitalización de los sistemas sanitarios ya es una realidad: impresión 3D, gemelos digitales aplicados a la medicina, consultas telemáticas o dispositivos médicos portátiles. En este contexto, la colaboración y compartición de datos médicos, siempre y cuando se garantice su protección, contribuye a impulsar la investigación e innovación en el sector. Es decir, las iniciativas de datos abiertos para la investigación médica estimulan este avance tecnológico en la salud.
Por todo ello, la Fundación FISABIO conjuntamente con el Centro de Investigación Príncipe Felipe en donde se ubica la plataforma que alberga BIMCV, se destaca como un ejemplo destacado al promover la apertura y compartición de datos en el campo de la medicina. A medida que avanza la era digital, es fundamental seguir fomentando la apertura de datos y promoviendo su uso responsable en la investigación médica, en beneficio de toda la sociedad.
En este post describimos paso a paso un ejercicio de ciencia de datos en el que tratamos de entrenar un modelo de deep learning con el objetivo de clasificar automáticamente imágenes médicas de personas sanas y enfermas.
El diagnóstico por radio-imagen existe desde hace muchos años en los hospitales de los países desarrollados, sin embargo, siempre ha existido una fuerte dependencia de personal altamente especializado. Desde el técnico que opera los instrumentos hasta el médico radiólogo que interpreta las imágenes. Con nuestras capacidades analíticas actuales, somos capaces de extraer medidas numéricas como el volumen, la dimensión, la forma y la tasa de crecimiento (entre otras) a partir del análisis de imagen. A lo largo de este post trataremos de explicarte, mediante un sencillo ejemplo, la potencia de los modelos de inteligencia artificial para ampliar las capacidades humanas en el campo de la medicina.
Este post explica el ejercicio práctico (sección Action) asociado al informe “Tecnologías emergentes y datos abiertos: introducción a la ciencia de datos aplicada al análisis de imagen”. Dicho informe introduce los conceptos fundamentales que permiten comprender cómo funciona el análisis de imagen, detallando los principales casos de aplicación en diversos sectores y resaltando el papel de los datos abiertos en su ejecución.
Proyectos previos
No podríamos haber preparado este ejercicio sin el trabajo y el esfuerzo previo de otros entusiastas de la ciencia de datos. A continuación, te dejamos una pequeña nota y las referencias a estos trabajos previos.
- Este ejercicio es una adaptación del proyecto original de Michael Blum sobre el desafío STOIC2021 - dissease-19 AI challenge. El proyecto original de Michael, partía de un conjunto de imágenes de pacientes con patología Covid-19, junto con otros pacientes sanos para hacer contraste.
- En una segunda aproximación, Olivier Gimenez utilizó un conjunto de datos similar al del proyecto original publicado en una competición de Kaggle. Este nuevo dataset (250 MB) era considerablemente más manejable que el original (280GB). El nuevo dataset contenía algo más de 1000 imágenes de pacientes sanos y enfermos. El código del proyecto de Olivier puede encontrarse en el siguiente repositorio.
Conjuntos de datos
En nuestro caso, inspirándonos en estos dos fantásticos proyectos previos, hemos construido un ejercicio didáctico apoyándonos en una serie de herramientas que facilitan la ejecución del código y la posibilidad de examinar los resultados de forma sencilla. El conjunto de datos original (de rayos-x de tórax) comprende 112.120 imágenes de rayos-X (vista frontal) de 30.805 pacientes únicos. Las imágenes se acompañan con las etiquetas asociadas de catorce enfermedades (donde cada imagen puede tener múltiples etiquetas), extraídas de los informes radiológicos asociados utilizando procesamiento de lenguaje natural (NLP). Partiendo del conjunto de imágenes médicas original hemos extraído (utilizando algunos scripts) una muestra más pequeña y acotada (tan solo personas sanas frente a personas con una sola patología) para facilitar este ejercicio. En particular, la patología escogida es el neumotórax.
Si quieres ampliar la información sobre el campo de procesamiento del lenguaje natural puedes consultar el siguiente informe que ya publicamos en su momento. Además, en el post 10 repositorios de datos públicos relacionados con la salud y el bienestar se cita al NIH como un ejemplo de fuente de datos sanitarios de calidad. En particular, nuestro conjunto de datos está disponible públicamente aquí.
Herramientas
Para la realización del tratamiento previo de los datos (entorno de trabajo, programación y redacción del mismo) se ha utilizado R (versión 4.1.2) y RStudio (2022-02-3). Los pequeños scripts de ayuda a la descarga y ordenación de ficheros se han escrito en Python 3.
Acompañando a este post, hemos creado un cuaderno de Jupyter con el que poder experimentar de forma interactiva a través de los diferentes fragmentos de código que van desarrollando nuestro ejemplo. El objetivo de este ejercicio es entrenar a un algoritmo para que sea capaz de clasificar automáticamente una imagen de una radiografía de pecho en dos categorías (persona enferma vs persona no-enferma). Para facilitar la ejecución del ejercicio por parte de los lectores que así lo deseen, hemos preparado el cuaderno de Jupyter en el entorno de Google Colab que contiene todos los elementos necesarios para reproducir el ejercicio paso a paso. Google Colab o Collaboratory es una herramienta gratuita de Google que te permite programar y ejecutar código en Python (y también en R) sin necesidad de instalar ningún software adicional. Es un servicio online y para usarlo tan solo necesitas tener una cuenta de Google.
Flujo lógico del análisis de datos
Nuestro cuaderno de Jupyter, realiza las siguientes actividades diferenciadas que podrás seguir en el propio documento interactivo cuándo lo vayas ejecutando sobre Google Colab.
- Instalación y carga de dependencias.
- Configuración del entorno de trabajo
- Descarga, carga y pre-procesamiento de datos necesarios (imágenes médicas) en el entorno de trabajo.
- Pre-visualización de las imágenes cargadas.
- Preparación de los datos para entrenamiento del algoritmo.
- Entrenamiento del modelo y resultados.
- Conclusiones del ejercicio.
A continuación, hacemos un repaso didáctico del ejercicio, enfocando nuestras explicaciones en aquellas actividades que son más relevantes respecto al ejercicio de análisis de datos:
- Descripción del análisis de datos y entrenamiento del modelo
- Modelización: creación del conjunto de imágenes de entrenamiento y entrenamiento del modelo
- Analisis del resultado del entrenamiento
- Conclusiones
Descripción del análisis de datos y entrenamiento del modelo
Los primeros pasos que encontraremos recorriendo el cuaderno de Jupyter son las actividades previas al análisis de imágenes propiamente dicho. Como en todos los procesos de análisis de datos, es necesario preparar el entorno de trabajo y cargar las librerías necesarias (dependencias) para ejecutar las diferentes funciones de análisis. El paquete de R más representativo de este conjunto de dependencias es Keras. En este artículo ya comentamos sobre el uso de Keras como framework de Deep Learning. Adicionalmente también son necesarios los siguientes paquetes: httr; tidyverse; reshape2;patchwork.
A continuación, debemos descargar a nuestro entorno el conjunto de imágenes (datos) con el que vamos a trabajar. Como hemos comentado previamente, las imágenes se encuentran en un almacenamiento remoto y solo las descargamos en Colab en el momento de analizarlas. Tras ejecutar las secciones de código que descargan y descomprimen los ficheros de trabajo que contienen las imágenes médicas encontraremos dos carpetas (No-finding y Pneumothorax) que contienen los datos de trabajo.

Una vez que disponemos de los datos de trabajo en Colab, debemos cargarlas en la memoria del entorno de ejecución. Para ello hemos creado una función que verás en el cuaderno denominada process_pix(). Esta función, va a buscar las imágenes a las carpetas anteriores y las carga en memoria, además de pasarlas a escala de grises y normalizarlas todas a un tamaño de 100x100 pixels. Para no exceder los recursos que nos proporciona de forma gratuita Google Colab, limitamos la cantidad de imágenes que cargamos en memoria a 1000 unidades. Es decir, el algoritmo va a ser entrenado con 1000 imágenes, entre las que va usar para entrenamiento y las que va a usar para la validación posterior.
Una vez tenemos las imágenes perfectamente clasificadas, formateadas y cargadas en memoria hacemos una visualización rápida para verificar que son correctas.Obtenemos los siguientes resultados:

Obviamente, a ojos de un observador no experto no se ven diferencias significativas que nos permitan extraer ninguna conclusión. En los siguientes pasos veremos cómo el modelo de inteligencia artificial sí que tiene mejor ojo clínico que nosotros.
Modelización
Creación del conjunto de imágenes de entrenamiento
Como comentamos en los pasos previos, disponemos de un conjunto de 1000 imágenes de partida cargadas en el entorno de trabajo. Hasta este momento, tenemos clasificadas (por un especialista en rayos-x) aquellas imágenes de pacientes que presentan indicios de neumotórax (en la ruta "./data/Pneumothorax") y aquellos pacientes sanos (en la ruta "./data/No-Finding")
El objetivo de este ejercicio es, justamente, demostrar la capacidad de un algoritmo de asistir al especialista en la clasificación (o detección de signos de enfermedad en la imagen de rayos-x). Para esto, tenemos que mezclar las imágenes, para conseguir un conjunto homogéneo que el algoritmo tendrá que analizar y clasificar valiéndose solo de sus características. El siguiente fragmento de código, asocia un identificador (1 para personas enfermas y 0 para personas sanas) para, posteriormente, tras el proceso de clasificación del algoritmo, poder verificar aquellas que el modelo ha clasificado de forma correcta o incorrecta.

Bien, ahora tenemos un conjunto “df” uniforme de 1000 imágenes mezcladas con pacientes sanos y enfermos. A continuación, dividimos en dos este conjunto original. El 80% del conjunto original, lo vamos a utilizar para entrenar el modelo. Esto es, el algoritmo utilizará las características de las imágenes para crear un modelo que permita concluir si una imagen se corresponde con el identificador 1 o 0. Por otro lado, el 20% restante de la mezcla homogénea la vamos a utilizar para comprobar si el modelo, una vez entrenado, es capaz de tomar una imagen cualquiera y asignarle el 1 o el 0 (enfermo, no enfermo).

Entrenamiento del modelo
Listo, solo nos queda configurar el modelo y entrenar con el anterior conjunto de datos.
Antes de entrenar, veréis unos fragmentos de código que sirven para configurar el modelo que vamos a entrenar. El modelo que vamos a entrenar es de tipo clasificador binario. Esto significa que es un modelo que es capaz de clasificar los datos (en nuestro caso imágenes) en dos categorías (en nuestro caso sano o enfermo). El modelo escogido se denomina CNN o Convolutional Neural Network. Su propio nombre ya nos indica que es un modelo de redes neuronales y por lo tanto cae dentro de la disciplina de Deep Learning o aprendizaje profundo. Estos modelos se basan en capas de características de los datos que se van haciendo más profundas a medida que la complejidad del modelo aumenta. Os recordamos que el término deep hace referencia, justamente, a la profundidad del número de capas mediante las cuales estos modelos aprenden.
Nota: los siguientes fragmentos de código son los más técnicos del post. La documentación introductoria se puede encontrar aquí, mientras que toda la documentación técnica sobre las funciones del modelo está accesible aquí.
Finalmente, tras la configuración del modelo, estamos en disposición de entrenar el modelo. Como comentamos, entrenamos con el 80% de las imágenes y validamos el resultado con el 20% restante.

Resultado del entrenamiento
Bien, ya hemos entrenado nuestro modelo. ¿Y ahora qué? Las siguientes gráficas nos proporcionan una visualización rápida sobre cómo se comporta el modelo sobre las imágenes que hemos reservado para validar. Básicamente, estas figuras vienen a representar (la del panel inferior) la capacidad del modelo de predecir la presencia (identificador 1) o ausencia (identificador 0) de enfermedad (en nuestro caso neumotórax). La conclusión es que cuando el modelo entrenado con las imágenes de entrenamiento (aquellas de las que se sabe el resultado 1 o 0) se aplica al 20% de las imágenes de las cuales no se sabe el resultado, el modelo acierta aproximadamente en el 85% (0.87309) de las ocasiones.

En efecto, cuando solicitamos la evaluación del modelo para saber qué tan bien clasifica enfermedades el resultado nos indica la capacidad de nuestro modelo recién entrenado para clasificar de forma correcta el 0.87309 de las imágenes de validación.
Hacemos ahora algunas predicciones sobre imágenes de pacientes. Es decir, una vez entrenado y validado el modelo, nos preguntamos cómo va a clasificar las imágenes que le vamos a dar ahora. Como sabemos "la verdad" (lo que se denomina el ground truth) sobre las imágenes, comparamos el resultado de la predicción con la verdad. Para comprobar los resultados de la predicción (que variarán en función del número de imágenes que se usen en el entrenamiento) se utiliza lo que en ciencia de datos se denomina la matriz de confusión. La matriz de confusión:
- coloca en la posición (1,1) los casos que SÍ tenían enfermedad y el modelo clasifica como "con enfermedad"
- coloca en la posición (2,2), los casos que NO tenían enfermedad y el modelo clasifica como "sin enfermedad"
Es decir, estas son las posiciones en las que el modelo "acierta" en su clasificación.
En las posiciones contrarias, es decir, la (1,2) y la (2,1) son las posiciones en las que el modelo se "equivoca". Así, la posición (1,2) son los resultados que el modelo clasifica como CON enfermedad y la realidad es que eran pacientes sanos. La posición (2,1), justo lo contrario.
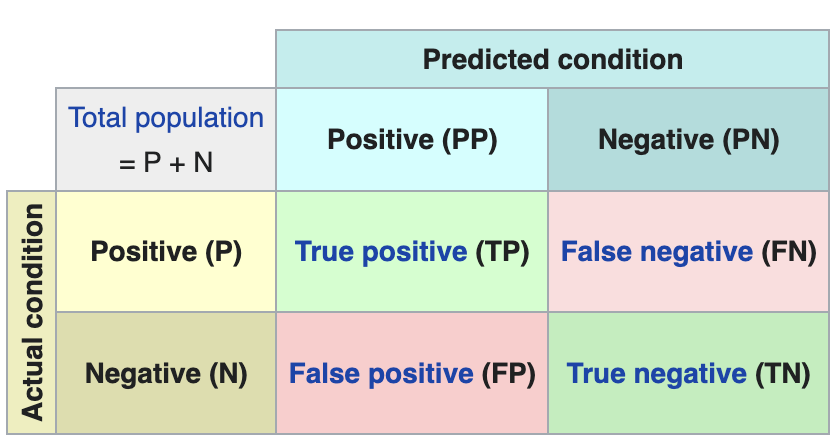
Ejemplo explicativo sobre cómo funciona la matriz de confusión. Fuente: wikipedia https://en.wikipedia.org/wiki/Confusion_matrix
En nuestro ejercicio, el modelo nos proporciona los siguientes resultados:


Es decir, 81 pacientes tenían esta enfermedad y el modelo los clasifica de forma correcta. De la misma forma, 91 pacientes estaban sanos y el modelo los clasifica, igualmente, de forma correcta. Sin embargo, el modelo clasifica cómo enfermos, 13 pacientes que estaban sanos. Y al contrario, el modelo clasifica cómo sanos a 12 pacientes que en realidad estaban enfermos. Cuando sumamos los aciertos del modelo 81+91 y lo dividimos sobre la muestra de validación total obtenemos el 87% de precisión del modelo.
Conclusiones
En este post te hemos guiado a través de un ejercicio didáctico que consiste en entrenar un modelo de inteligencia artificial para realizar clasificaciones de imágenes de radiografías de pecho con el objetivo de determinar automáticamente si una persona está enferma o sana. Por sencillez, hemos escogido pacientes sanos y pacientes que presentan un neumotórax (solo dos categorías) previamente diagnosticados por un médico. El viaje que hemos realizado nos ofrece una idea de las actividades y las tecnologías involucradas en el análisis automatizado de imágenes mediante inteligencia artificial. El resultado del entrenamiento nos ofrece un sistema de clasificación razonable para el screening automático con un 87% de precisión en sus resultados. Los algoritmos y las tecnologías avanzadas de análisis de imagen son y cada vez más serán, un complemento indispensable en múltiples ámbitos y sectores como, por ejemplo, la medicina. En los próximos años veremos cómo se consolidan los sistemas que, de forma natural, combinan habilidades de humanos y máquinas en procesos costosos, complejos o peligrosos. Los médicos y otros trabajadores verán aumentadas y reforzadas sus capacidades gracias a la inteligencia artificial. La combinación de fuerzas entre máquinas y humanos nos permitirá alcanzar cotas de precisión y eficiencia nunca vistas hasta la fecha. Esperamos que con este ejercicio os hayamos ayudado a entender un poco más cómo funcionan estas tecnologías. No olvides completar tu aprendizaje con el resto de materiales que acompañan este post.
Contenido elaborado por Alejandro Alija, experto en Transformación Digital. Los contenidos y los puntos de vista reflejados en esta publicación son responsabilidad exclusiva de su autor.
La salud es uno de los campos de desarrollo prioritario en este siglo. La mayoría de los analistas coinciden en que la gestión de la salud - desde todas las ópticas posibles - cambiará de forma radical en los próximos años. El análisis de los datos de salud marcará el camino a seguir en las épocas venideras.
La esperanza de vida de los países desarrollados aumenta a medida que avanza el siglo. En los últimos veinte años, la esperanza de vida (EV) de muchos países desarrollados ha superado la barrera de los 80 años de media. Japón, España, Suiza, Singapur, entre otros, se sitúan ya por encima de los 83 años de esperanza de vida y la tendencia continúa con una tasa de crecimiento continuado.
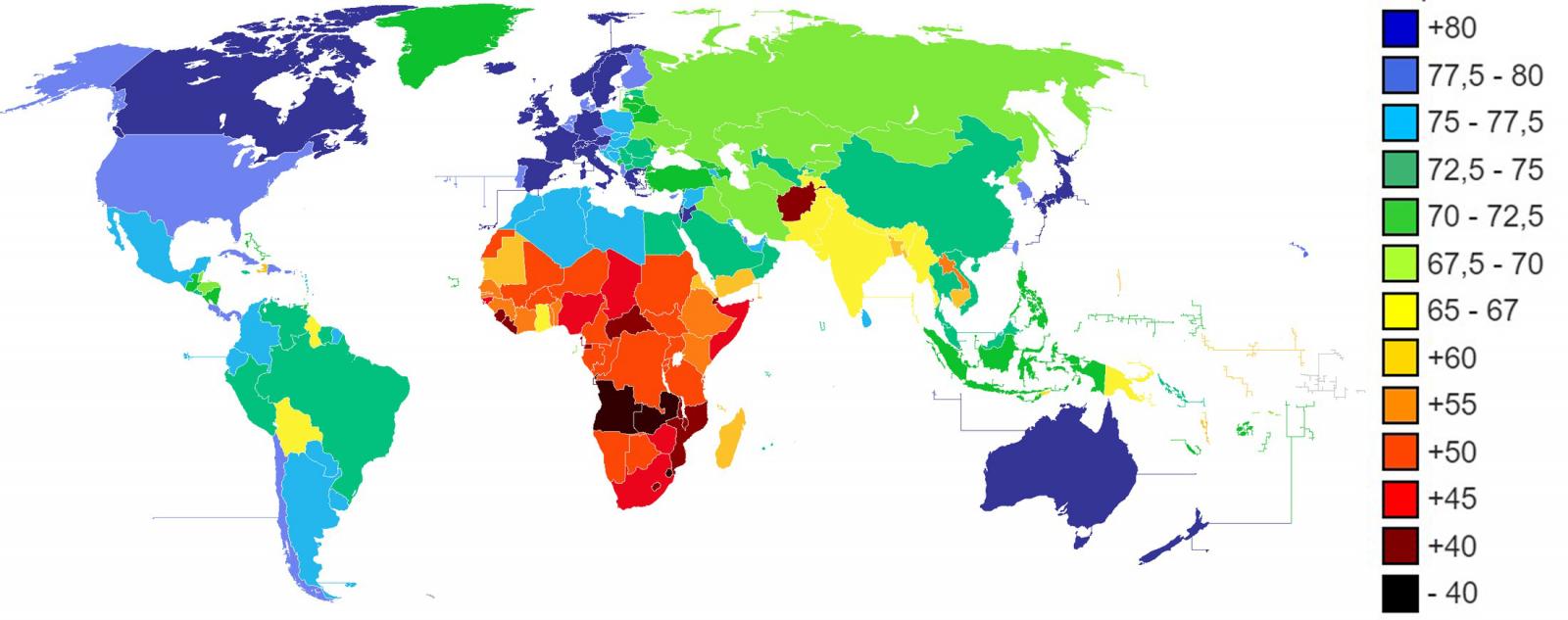
Figura 1. Esperanza de vida en años según el CIA World Factbook 2013.
Valga esta introducción sobre la esperanza de vida para motivar el tema central de este artículo. A medida que envejecemos, las enfermedades que nos afectan van evolucionando. Una mayor esperanza de vida no significa necesariamente una mejor calidad de vida en la etapa adulta y anciana. Para vivir más años es necesario desarrollar un mejor cuidado de la salud. Las sociedades modernas necesitan realizar una transición satisfactoria desde el tratamiento a la prevención. Es decir: Prevenir antes que curar.
Pero prevenir pasa, necesariamente, por conocer mejor los riesgos y anticiparse a futuras complicaciones. El análisis de los datos relacionados con nuestra salud es de capital importancia para afrontar esta transición. No son pocas las tareas y acciones necesarias antes de llegar a establecer estrategias continuadas de análisis de datos de salud.
Los datos relacionados con la salud son, por naturaleza, datos de carácter sensible. Los datos personales de salud, tienen implicaciones directas sobre nuestras relaciones laborales y personales y pueden llegar a impactar de forma muy notoria sobre nuestra economía -a nivel personal como de sociedad-. Los desafíos a los que se enfrenta el análisis de datos de salud son, entre otros:
-
Generación de conjuntos de datos (datasets) públicos.
-
Mecanismos estándar de anonimización de datos de salud.
-
Herramientas de recolección de datos de salud en tiempo real.
-
Modelos de datos de salud consensuados por la comunidad científica.
-
Herramientas de análisis de datos de salud preparadas para grandes ingestas y altos volúmenes de datos.
-
Perfiles especialistas, tanto conocedores del dominio de la salud como científicos de datos especializados en este campo (datos semi-estructurados y tecnologías semánticas).
Transformación digital del sector salud
La transformación digital del sector salud representa uno de los mayores desafíos para instituciones y sistemas de salud tanto públicos como privados. Buena parte de los centros hospitalarios de países desarrollados han comenzado a digitalizar parte de los datos más importantes en relación a nuestra salud. Especialmente, aquellos datos registrados en las visitas presenciales a centros de salud y hospitales. La historia clínica digital o EHR por sus siglas en inglés (Electronic Health Records) así como las pruebas diagnósticas (por ejemplo, imágen médica o análisis clínicos) son los registros con mayor grado de digitalización. Si bien es cierto que el grado de digitalización de estos ejemplos puede llegar a ser alto, la forma en la que se ha planteado es diferente por países y sistemas. Convertir en información digital las históricas clínicas analógicas añade muy poco valor comparado con el esfuerzo y la inversión necesaria. Sin embargo, afrontar la digitalización de las historias clínicas con el foco puesto en el posterior análisis inteligente de los datos puede suponer una revolución con un impacto incalculable. Por ejemplo, la implementación de ontología especialmente diseñada para el dominio médico como SNOMED-CT cambia de forma radical la explotación futura de los datos médicos y habilita una capa superior de inteligencia apoyada en la futura Inteligencia Artificial como asistente de los médicos y enfermeras del futuro.
Algunos repositorios públicos
Existen diferentes repositorios donde encontrar conjuntos de datos abiertos con caracter de salud. La mayor parte de datos disponibles consistente en estadísticas relacionadas con indicadores de salud. Sin embargo, existen repositorios más especializados donde es posible encontrar conjuntos de datos sobre los cuales realizar analítica de datos avanzada.
Por ejemplo, los sistemas de salud de EEUU y Reino Unido respectivamente, publican sus datos de salud en los repositorios:
Otras organizaciones multi-país, como la Organización Mundial de la salud (OMS) o la ONG Unicef también disponen de repositorios de datos abiertos:
-
UNICEF ofrece estadísticas sobre la situación de las mujeres y los niños en todo el mundo.
-
World Health Organization ofrece estadísticas mundiales sobre el hambre, la salud y las enfermedades.
Más allá de los datos estadísticos, el sitio web especializado en ciencia de datos Kaggle convoca periódicamente competiciones abiertas en las que los equipos pueden participar para resolver desafíos basados en datos. Por ejemplo, en una de las competiciones de Kaggle, el desafío consistía en predecir reingresos hospitalarios por diabetes. Para resolver el desafío, se disponía de un conjunto de datos (debidamente anonimizado) compuesto por 65 registros de pacientes con diabetes y 50 campos que incluyen información de: género, edad, peso, etc.
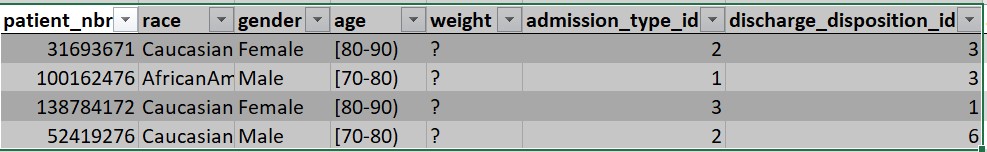
Figura 2. Extracto del conjunto de datos disponible para el desafío de diabetes.
En resumen, el análisis sistemático de datos de salud abre las puertas de la medicina predictiva. Para habilitar tecnologías que asistan a los profesionales sanitarios del futuro es necesario construir estrategias de datos sostenibles, escalables y duraderas. Recolectar, almacenar, modelar y analizar (RAMA) datos de salud es la clave para un futuro en el que el cuidado de la salud sea algo más que un mero contacto asistencial con los pacientes.
Contenido elaborado por Alejandro Alija,experto en Transformación Digital e Innovación.
Los contenidos y los puntos de vista reflejados en esta publicación son responsabilidad exclusiva de su autor.